Tech Blog
[프로젝트] Crop Doctor: 인공지능 기반 웹 서비스 개발 회고 본문
5주에 걸쳐 인공지능 기반의 웹 서비스를 개발하는 팀 프로젝트를 수행하였다. 이 프로젝트를 통해 인공지능을 활용한 기능이 메인서비스인 기획, 그에 대한 인공지능 모델을 직접 학습, 웹 서비스 개발 맟 배포까지 기획 부터 배포까지의 전반적인 과정을 경험할 수 있었다.
먼저, 기획 주제는 다음과 같이 선정하였다.
작물의 질병을 진단해주는 웹 서비스: Crop Doctor
🐈⬛ github
작물의 질병을 진단해주고 해결책을 제시해주는 웹 서비스이다. 부가 기능으로 작물 성장일지 커뮤니티와 질병이 있는 작물에게 뿌릴 수 있는 영양제 정보를 담은 페이지까지 개발하는 것으로 기획하였다. 인공지능, 프론트엔드, 백엔드 포지션 중에 인공지능과 백엔드 파트를 담당하여 개발을 진행하였다.
1. 인공지능 모델 학습
🌱 언어 & 프레임워크: python & pytorch
🌱 개발 환경: Lunux Server Jupyter Lab
모델 학습 과정
1.1 데이터 수집 및 적재
🗃️ 사용 데이터셋

선정한 데이터셋은 기획한 서비스인 작물의 질병 진단 모델을 학습할 수 있는 AIHub의 노지작물 질병 진단 이미지이다.
💡 용량 문제
그런데 위의 용량 부분에서 볼 수 있듯이 용량이 609.46GB였다. 하지만 프로젝트에서 제공받은 VM의 디스크 용량은 300GB까지여서 데이터를 줄여야했다.

데이터를 살펴보니 증강 데이터가 용량을 많이 차지하고 있었다. 물리적으로 데이터를 변형시켜 제공해주었으며, 대부분 증강 데이터의 용량이 원본 데이터보다 훨씬 컸다. 그래서 증강 데이터는 삭제하고, 모델 학습 시 transform 함수를 사용하여 증강을 시도하기로 하였다. 최종적으로 증강 데이터를 제외한 총 247GB의 데이터를 팀원들과 나누어 수집하였다.
💡 VM 적재 및 압축 해제 문제
로컬에 다운받은 데이터셋을 VM에 적재하는 과정에서도 조금의 이슈가 있었다. 용량이 15GB가 넘어가는 데이터는 제대로 올라가지 않고 손상되어 올라갔다.
BadZipFile: File is not a zip file손상된 파일을 리눅스의 unzip 명령어로 압축해제 할 때 위와 같은 오류가 발생했다. 처음에는 원인을 알지 못해서 손상된 zip 파일 복구를 시도하였지만, 원본 zip 파일과 VM에 올라온 zip 파일의 용량이 다르다는 것을 확인하고 제대로 올라오지 않음을 알게되었다.
위의 두 가지의 이슈가 있었고, 다음과 같은 과정으로 데이터 수집 및 VM 업로드하였다.
1) 1TB 하드에 609GB 다운로드 -> VM 업로드 -> zip 압축해제 안 됨
한번에 올리니 모든 파일 손상, 디스크 용량 300GB임을 전달받음
2) 증강 데이터 제외 247GB 를 4명이 나누어 다운 -> VM에 zip 파일 하나씩 업로드 -> 일부 zip 파일만 압축해제 됨
용량이 큰 데이터는 한개씩 올리더라도 여전히 파일이 손상되는 이슈가 있었기 때문에 손상되지 않은 데이터들로만 모델 학습을 진행하기로 함. 그래서 최종적으로 약 150GB의 데이터를 업로드하는데 성공
데이터 수집 및 업로드에 거의 1주의 기간이 걸렸다. 총 5주간의 기간 중 2주차 중반까지는 모델 학습을 할 수 없었다. 데이터의 용량이 커 다운로드및 업로드를 할 때의 시간 때문에 더 오래 걸렸던 것 같다. 인공지능 모델 학습을 위해서는 대용량의 데이터가 필요하고, 그에 따른 고성능 장비가 요구되기 때문에 규모가 작은 프로젝트에서는 해당 환경에 맞게 맞춰나가야 한다는 점과, 그에 대한 해결능력을 배웠다.
1.2 EDA / 전처리
Data Analysis blog - [cropdoctor] 1. 노지작물 데이터 EDA
🪴 [cropdoctor] 인공지능 기반 웹서비스 개발 프로젝트 1. EDA 사용 데이터셋 노지 작물 질병 진단 이미지 인공지능 기반의 웹 서비스 개발 프로젝트에서 작물의 질병을 진단 주제를 선정했습니다.
ag-su.github.io
VM에 업로드한 데이터에 대한 EDA를 진행했다. 데이터는 레이블 데이터, 이미지 데이터, 총 2가지로 이루어져있다.

레이블 데이터의 형태를 먼저 살펴봤다. 이미지 데이터 하나당 하나의 json 파일로, 위와 같은 형태를 띄고있었다. 레이블 데이터를 사용할 때마다 파일을 하나씩 불러오는 것은 비효율적이라는 생각이 들어, 하나의 딕셔너리 파일로 모으는 전처리를 진행하였다. 그 중에서도 필요한 필드인 질병, 작물, 바운딩박스, 이미지 크기만 담는 것으로 설계 하였다. 처리된 딕셔너리는 다음과 같은 형태를 띈다.
{image: {disease, crop, points, (width, height)}, …}

이미지 데이터는 위와 같이 바운딩박스를 포함하여 9장을 랜덤으로 뽑아 살펴보았습니다. 기본적으로 Image classification 학습을 할 수 있으며, 바운딩박스가 제공되므로 Object Detection 모델로도 학습을 해볼 수 있을 것으로 보입니다.
💡 데이터 불균형 문제

앞서 처리한 레이블 데이터의 딕셔너리를 사용하여 클래스 별 개수를 뽑아봤다. 대체적으로 정상 데이터의 개수 대비 질병 데이터의 개수가 현저히 적었다.
랜덤 샘플링
따라서 랜덤 샘플링을 진행하기로 했다. 불균형 문제 뿐만 아니라, 용량이 커서 VM이 자주 다운되는 현상이 있었기 때문에 각 정상 데이터에서 1000장씩 랜덤으로 샘플링하는 방법을 선택하였다.

샘플링 후에도 거의 모든 작물에 불균형이 남아있었다. 이는 학습 시 WCE(weighted cross entropy )를 사용하여 보완하기로 하였다.
EDA를 통해 데이터를 탐색하는 과정을 통해 데이터 처리와, 모델 학습을 어떻게 진행해야 할지 설계할 수 있었다. 특히, 이미지 데이터의 예시 사진 확인과 레이블 데이터의 구성, 데이터 불균형 이슈 발견으로, 모델 학습 방법이나 모델 학습 시 데이터의 처리에 대한 감을 얻을 수 있었다.
1.3 모델 학습 및 평가
1) AIHub 샘플 모델
[cropdoctor] 2. tar 확장자 모델 docker image 불러오기
Data Analysis blog - [cropdoctor] 2. tar 확장자 모델 docker image 불러오기
이미지로 확인해보기 lst_nums = random.sample(range(len(lst_img)), 9) dict_name = {0: 0, 1: 7, 2: 8} fig = plt.figure(figsize=(13, 13)) axes = fig.subplots(3, 3).flatten() for i, num in enumerate(lst_nums): img_name = lst_img[num] img = Image.open(
ag-su.github.io
처음으로, AIHub에서 제공하는 sample 모델을 불러와 어느 정도의 성능이 나오는지 확인하였다.
💡 tar 확장자 Docker Image 불러오기
docker load -i 73.tarCannot connect to the Docker daemon at unix:///var/run/docker.sock. Is the docker daemon running?
sudo systemctl status dockerSystem has not been booted with systemd as init system (PID 1). Can’t operate. Failed to connect to bus: Host is down
VM에서 tar 확장자를 docker image로 로드하려고 시도했지만, 위와 같은 오류가 났다. 데몬이 실행되고 있는지 확인해본 두번째 명령어에서의 오류는 systemd가 init 시스템으로 설정되어 있지 않거나, 호스트가 다운되었기 때문에 발생하는 것이라고 한다. 결론적으로는 서버 운영진 측에 문의한 결과 제공받은 VM에서 docker를 지원하지 않는다고 전달받았고, 로컬에서 모델을 받아와야 했다.
로컬에서 docker image를 받고, VM에 업로드하는 과정은 위의 [cropdoctor] 2. tar 확장자 모델 docker image 불러오기 글에서 자세히 확인할 수 있습니다. 대략적인 과정은 다음과 같습니다.
로컬에서 도커 이미지 불러오기 -> 도커 컨테이너 실행 -> 도커 컨테이너에서 로컬로 복사 -> 로컬에서 VM으로 업로드

모델의 구조를 확인해봤을 때 마지막 FC레이어를 보면, (1, 3)임을 볼 수 있다.

docker 파일 내 config 함수를 확인해보니, 04 작물에 대하여 정상/질병1/질병2 와 같이 분류하는 모델이었고, 전체 작물에 대하여 분류하는 모델은 아니였다.
따라서 04 작물인 애호박 데이터로 모델의 성능을 평가하여 평가지표의 수치를 참고하기로 하였다.

평가지표를 계산해보니 위와 같은 결과가 나왔다. 성능이 우수하지는 않은 그냥 정말 샘플 모델이었던 것 같다. 참고만 하는 걸로..
2) Image Classification (MobilenetV2)
[cropdoctor] 3. 작물 질병 진단 모델 학습 및 평가 (mobilenetV2)
Data Analysis blog - [cropdoctor] 3. 작물 질병 진단 모델 학습 및 평가 (mobilenetV2)
🪴 [cropdoctor] 인공지능 기반 웹서비스 개발 프로젝트 3. 작물 질병 진단 모델 학습 및 평가 (mobilenetV2) import pickle import os from tqdm import tqdm import random import time from PIL import Image, ImageDraw, ImageFile import
ag-su.github.io
두번째로는, 가장 기본적인 학습 방법 Image Classification 모델 학습이다. 전이학습을 진행하며, 사전학습 모델은 MobileNet V2 를 사용하였다. MobileNet V2 는 작은 모델 크기와 낮은 연산 요구를 유지하면서도 정확한 예측 결과를 얻을 수 있으므로, 프로젝트에서 제공받은 VM의 성능에서 무리 없이 돌아가게 하면서도, 높은 분류 성능을 제공해주므로 해당 모델을 선정하였다.
학습 모델의 구조는 다음과 같다.

input 224x224x3로 설정하였고, output은 클래스의 개수인 1x20으로 재 설정하였다.
데이터의 폴더구조는 다음과 같다.

딥러닝 프레임워크는 pytorch를 사용하였고, training, validation안의 image_class 폴더 안에 클래스 별 폴더로 나누어져 있는 폴더구조로, ImageFolder와 DataLoader 함수를 사용하여 데이터를 불러왔다.
💡 세션 끊김 문제
이미지 데이터다 보니 학습 시간이 오래 걸렸다. 1 epoch당 1시간 정도였기에 10 epoch을 돌릴 때는 10시간이 넘게 걸렸다. 하지만 그냥 모델 학습을 돌리면, 세션이 10시간 동안 살아있지 못 하기 때문에 서버가 재부팅되지 않는 이상 학습 중인 세션이 종료되지 않을 수 있는 tmux를 사용하여 모델학습을 진행하였다.
💡 데이터 불균형 문제
▪️ WCE (Weighted Cross Entropy, 가중 교차 엔트로피)
가중 교차 엔트로피(Weighted Cross Entropy)는 분류 문제에서 사용되는 손실 함수이다. 클래스별로 다른 가중치를 적용하여 모델의 학습을 조정하는 데 사용되는 기법이다. 일반적인 교차 엔트로피 손실 함수는 모든 클래스를 동등하게 취급하지만, 실제로는 데이터셋에 클래스 불균형이 존재하거나 특정 클래스의 중요도가 다를 수 있다. 이 때 클래스별로 다른 가중치를 적용하는 가중 교차 엔트로피를 사용할 수 있다.
nSamples = [1000, 973, 915, 1000, 470, 227, 1000, 802, 458, 1000, 543, 484, 1000, 538, 854, 1000, 216, 1001, 208, 226]
normedWeights = [1 - (x / sum(nSamples)) for x in nSamples]
normedWeights = torch.FloatTensor(normedWeights).to(device)
criterion = nn.CrossEntropyLoss(weight=normedWeights)위와 같이 클래스 별 개수에 따라 가중치를 설정하였다. 클래스의 개수가 적으면 더 높은 가중치를 가지며, 클래스 개수가 많을 수록 낮은 가중치를 가지게 함으로써 데이터 불균형 문제 보완을 시도하였다. (결론적으로 효과를 보지는 못 했다.)
▪️ 데이터 증강
train_transform = transforms.Compose([
transforms.RandomRotation(degrees=30),
transforms.RandomResizedCrop(size=(224, 224)),
transforms.RandomHorizontalFlip(p=0.5),
transforms.Resize((224, 224)),
transforms.ToTensor(),
transforms.Normalize(mean=[0.485, 0.456, 0.406], std=[0.229, 0.224, 0.225])
])
val_transform = transforms.Compose([ # 이미지 크기, 정규화만 진행
transforms.Resize((224, 224)),
transforms.ToTensor(),
transforms.Normalize(mean=[0.485, 0.456, 0.406], std=[0.229, 0.224, 0.225])
])앞서 랜덤샘플링으로 데이터의 수가 약 8만장에서 약 1만5천장으로 대폭 줄었다. transforms.Compose 함수를 사용한 데이터 증강으로 다양한 데이터셋을 학습할 수 있도록 하였다.
평가 지표 결과
(1) batch size: 64, learning_rate: 0.001, epoch: 10

(2) batch size: 64, learning_rate: 0.001, epoch: 10 + WCE, Transform

aihub의 샘플 모델과 비교했을 때 결과가 높게 나온 것으로 보인다. 하지만 데이터 증강, WCE를 추가하여 학습을 진행하여 데이터의 다양성과 불균형을 보완한 두번째 실험에서 크게 성능이 올라가지는 않았다.
epoch을 10보다 크게 설정하여 학습하지 못한 점과, 이외의 다른 하이퍼 파라미터를 변경하여 실험해보지 못한 점이 아쉬웠다.
3) Object Detection (Yolo V5)
[cropdoctor] 4. 작물 질병 데이터셋으로 커스텀 YOLO 모델 학습
Data Analysis blog - [cropdoctor] 4. 작물 질병 데이터셋으로 커스텀 YOLO 모델 학습
학습을 위해 생성한 파일 정리 1) images 폴더 안에 전체 이미지 파일 2) labels 폴더 안에 이미지 파일에 대응되는 레이블 정보 txt 파일 3) 전체 이미지 파일의 경로가 모두 적힌 txt 파일 4) 클래스 이
ag-su.github.io
다음은 Object Detection 방법 중 yolo v5 모델로 학습을 진행하였다.
데이터의 폴더구조는 다음과 같다.

앞서 보았던 image classification의 폴더구조와 다르게 전체 image를 하나의 폴더에 모으고, label 정보 (클래스, 바운딩박스 좌표)가 들어가있는 폴더를 따로 생성해주어야 했다. 여기서 바운딩박스는
원본 bbox: (x_min, y_min, x_max, y_max) - 좌상단, 우하단 꼭짓점 좌표
yolo bbox: (x_center, y_center, width, height)
와 같이 변형을 해주어 입력되어야 한다.
최종적으로, yolo 학습을 위해 생성한 파일들은 다음과 같다.
1) images 폴더 안에 전체 이미지 파일
2) labels 폴더 안에 이미지 파일에 대응되는 레이블 정보 txt 파일
3) 전체 이미지 파일의 경로가 모두 적힌 txt 파일
4) 클래스 이름, 개수, 3번에서 생성한 txt 파일 경로를 포함한 yml 파일
평가 지표 결과

실제와 예측 이미지를 비교한 사진입니다. 위의 도출된 결과 사진을 보면, pred의 bbox가 오히려 잎을 더 잘 예측한 것으로 보인다.

20 에폭 동안 이상적으로 평가지표는 올라가고 loss값은 줄어들지만, validation의 obj_loss값은 증가하는 추이를 보인다. 위의 3가지의 curve 그래프나, 이 tensorboard로 미루어 보아, 20 에폭으로는 학습이 덜 된 것 같다. epoch을 100 이상으로 설정하고 돌리면 더 괜찮은 결과를 보일 수 있을 것이라 예상된다.
메인기능 미리 보기

시간의 여유가 있었다면, 더 많은 모델 학습과 실험을 통해 결정하고 싶은 마음이 있었지만, image classification과 object detection 모델 학습을 비교 학습해봤다는 점에 의의를 두려고 한다. 최종적으로는 Image classification 방법의 mobilenet V2 로 전이학습 한 모델을 선정하여 웹서비스에 적용하였다. 작물의 사진을 넣으면, 우측에 진단 결과를 보여주는 흐름이다.
2. 백엔드
🌱 언어 / 프레임워크
- 서비스 api: TypeScript / Nest.js
- 인공지능 모델 예측 api: Python / FastAPI
🌱 데이터베이스: AWS RDS
* 폴더 구조

폴더구조는 위와 같다. 기능 별로 폴더를 나누고, 그 안에서 entities, repositories, 폴더와 controller, module, service 파일을 각각 생성하여 개발을 진행하였다.
1) 데이터베이스 설계

웹 서비스의 주요 기능인 질병진단-해결책, 영양제, 커뮤니티에서 필요한 테이블을 설계하였다. 유저와 해결책, 영양제와 카테고리와 같은 다대다 관계 등의 데이터 간의 관계를 표현하고 조인 연산을 사용하여 복잡한 쿼리를 수행하기 용이하므로, 관계형 데이터베이스를 선택하였다.
2) API 개발
▪️ 영양제/카테고리 CRUD
💡 이미지 업로드
영양제, 카테고리에 대한 CRUD 를 작성하였고, 그 중에서 기억에 남는 기능은 이미지 업로드이다. 이미지 업로드의 저장소는 AWS S3, 패키지는 multerS3를 사용하였다. 코드는 다음과 같이 작성했다.
utils/multer.options.ts
const s3Client = new S3Client({
region: process.env.AWS_S3_REGION,
credentials: {
accessKeyId: process.env.AWS_S3_ACCESS_KEY_ID,
secretAccessKey: process.env.AWS_S3_SECRET_ACCESSKEY,
},
});
export const multerOptions = (dirName: string): MulterOptions => {
const options = {
storage: multerS3({
s3: s3Client,
bucket: process.env.AWS_S3_BUCKET_NAME,
contentType: multerS3.AUTO_CONTENT_TYPE,
metadata: (req, file, cb) => {
cb(null, { fieldName: file.fieldname });
},
key: (req, file, cb) => {
const uploadDirectory = dirName;
cb(null, `${uploadDirectory}/${Date.now()}_${file.originalname}`);
},
}),
};
return options;
};먼저 multer 의 option을 설정해주는 multerOptions 함수는 utils 폴더에 따로 빼서 파일을 생성하였다. 여기서 aws s3 의 region, 발급받은 access key와 secret access key를 설정하여 s3Client 객체를 만들고, multerOptions 함수에서 s3Client 객체, bucket 이름 등을 설정한다. 또, 어느 디렉토리에 이름을 어떻게 저장할지도 설정한다.
위의 코드 같은 경우에는 파라미터에서 dirName 을 받아서 디렉토리를 설정하였다. 이미지 업로드는 유저 프로필, 영양제 추가, 질병 진단에서 사용되므로, dirName에 user, tonic, crop 을 적어 해당 폴더에 위치하도록 코드를 작성했다.
admin.controller.ts
@Post('/tonics')
@UseGuards(AdminAuthGuard)
@UseInterceptors(FileInterceptor('image', multerOptions('tonic')))
async createTonic(
@UploadedFile() file,
@Body() createTonicDto: CreateTonicDto,
) {
return await this.adminService.createTonic(file.location, createTonicDto);
}그 다음, 이미지 업로드를 사용하는 controller에서 @UseInterceptors 데코레이더 안에 FileInterceptor 함수로 첫번째 인자에는 요청받는 이미지의 필드명, 두번째 인자에는 작성해놓은 multerOptions 함수를 넣었다. 그럼 @UploadedFile() 데코레이더에서 file이라는 변수 안에 이미지 파일을 성공적으로 받을 수 있다.
▪️ 해결책 CRD
💡 유저 체크
solution.controller.ts
@Post('')
@UseGuards(AuthGuard())
async createUserSolution(
@GetUser() user: User,
@Body() createUserSolutionDto: CreateUsersolutionDto,
) {
// 요청: header token, solutionId, image, resolvedAt
// 응답: 저장된 해결책
return await this.solutionsService.createUserSolution(
user.id,
createUserSolutionDto,
);
}질병 진단을 하고, 그에 대한 해결책이 나오면 로그인 한 유저는 마이페이지에 저장을 할 수 있다. 따라서 해결책의 CRD는 유저 권한 체크를 시도해야 했다. 위와 같이 @UseGards 데코레이터에 AuthGaurd() 함수를 넣고, createUserSolution 함수의 인자에 @GetUser() 데코레이터로 해당하는 유저객체를 받아올 수 있었다.
▪️ 메인 기능: 작물 질병 진단
💡 nestjs 백엔드 서버와 fastapi 인공지능 서버의 통신
* 폴더 구조

인공지능 파트의 폴더구조는 위와 같다. main.py에 api 코드를 작성하고, 이미지 전처리 함수와 모델 예측 함수는 따로 funcs 폴더에서 작성하였다.
main.py
@app.post("/predict")
async def predict(image_key: str):
endpoint_url = os.getenv('endpoint_url')
access_key = os.getenv('aws_access_key_id')
secret_key = os.getenv('aws_secret_access_key')
s3 = boto3.client('s3', endpoint_url=endpoint_url, aws_access_key_id=access_key,
aws_secret_access_key=secret_key)
# S3 객체 다운로드
response = s3.get_object(Bucket='cropdoctor', Key=image_key)
img_data = response['Body'].read()
preprocessed_image = image_preprocessing(img_data)
probability, solution_id = get_prediction(preprocessed_image)
return JSONResponse({"solutionId": solution_id, "probability": probability})s3에 업로드된 이미지의 주소를 가져와서 이미지를 전처리 하는 것은 불가하였다. 따라서 백엔드에서 이미지 주소가 아닌 이미지 키를 요청하면, ai 서버에서 s3 버킷에 직접 접근하여 s3객체를 다운받아 이미지를 가져와서 사용해야했다. 최종적으로는 예측값인 solutionId와, 예측 확률 probability를 응답으로 전송되었다.
도식화 한 그림으로 살펴보겠다.
인공지능 모델 예측 흐름

백엔드 서버에서 인공지능 서버로 image_key 와 함께 POST 요청을 보내면 ai 서버에서 모델 예측을 진행하여 나온 결과값 prediction과 probability를 json으로 응답한다. 그 이후에는 백엔드 서버에서 DB의 solution 테이블에 접근하여 prediction값 (solution_id)에 맞는 해결책 내용을 불러와 클라이언트 측에 응답을 보내게 된다.
3) 배포
배포는 3개의 파트에 대하여 다음과 같이 진행하였다.
프론트: yarn build로 builld 폴더 생성 후, nginx에서 설정
백: pm2
인공지능: gunicorn
전체적인 서비스 아키텍처를 살펴보면, 다음과 같다.
▪️ 서비스 아키텍처

3. 서비스 시연
3.1 랜딩 페이지

3.2 메인기능: 작물 질병 진단
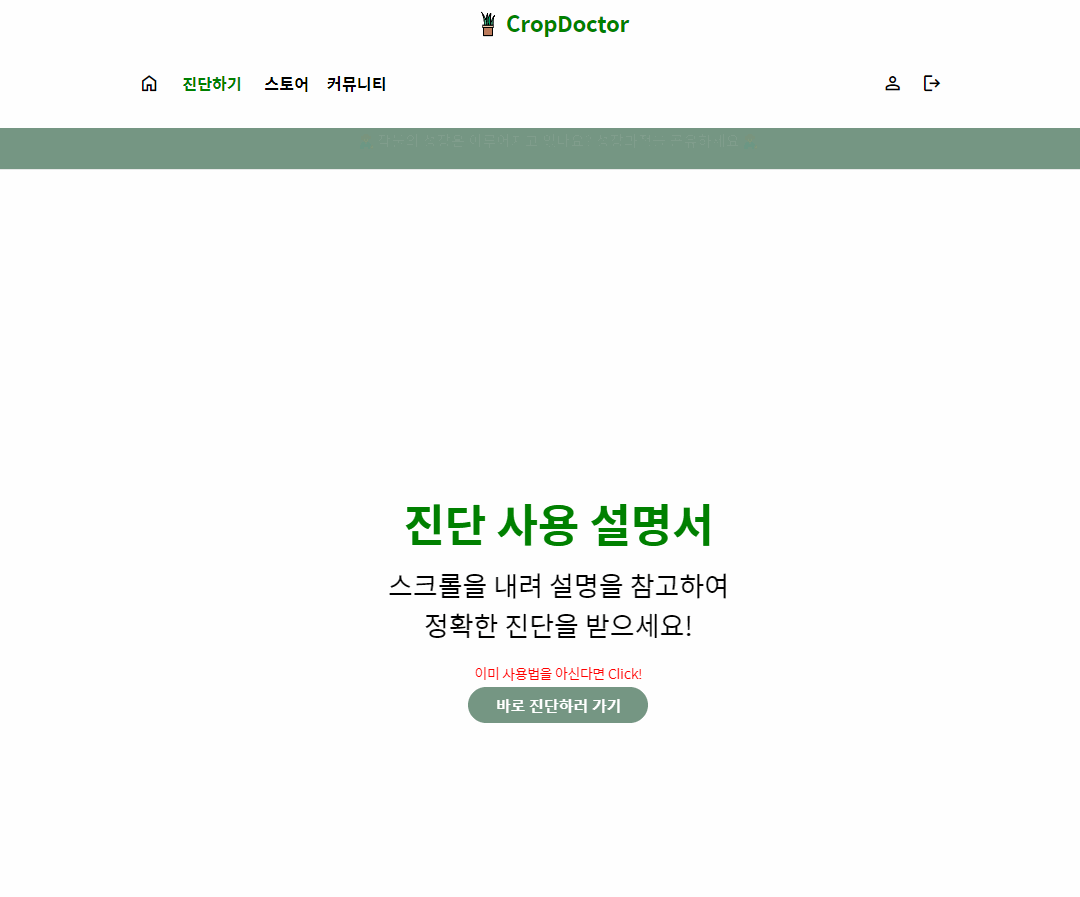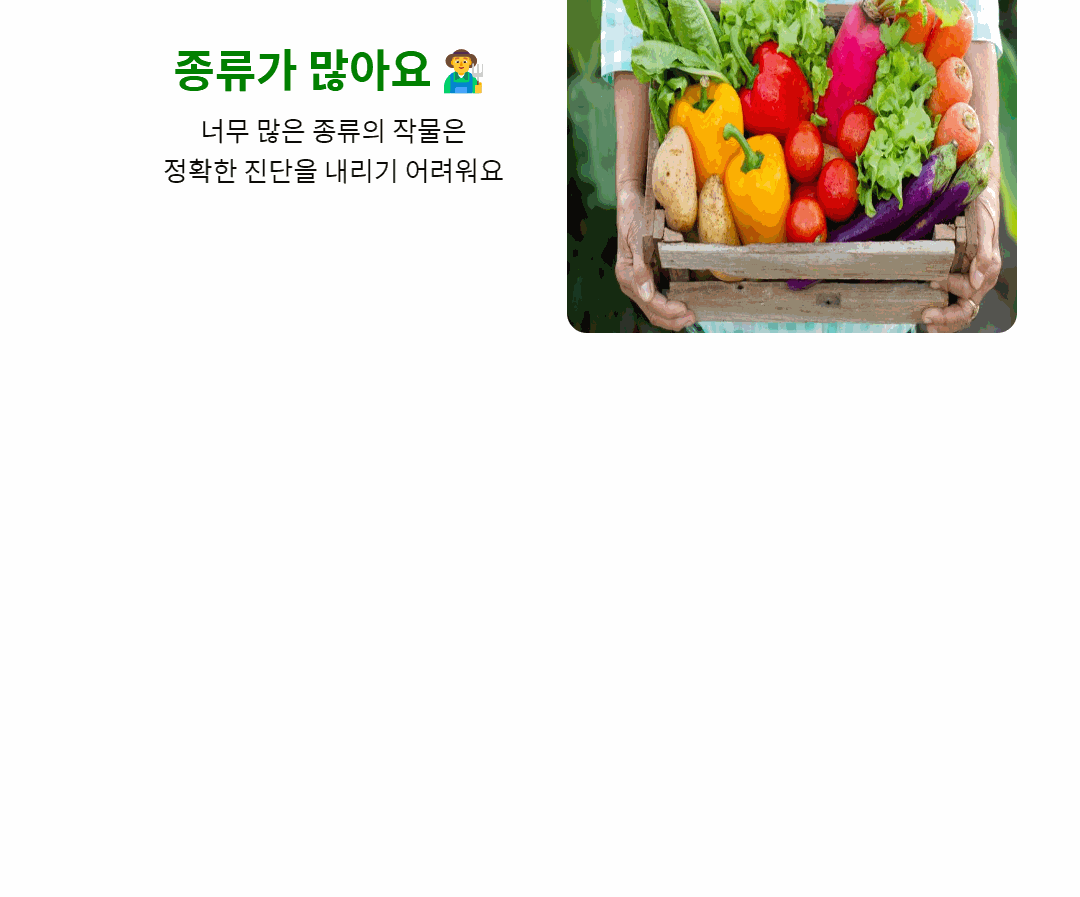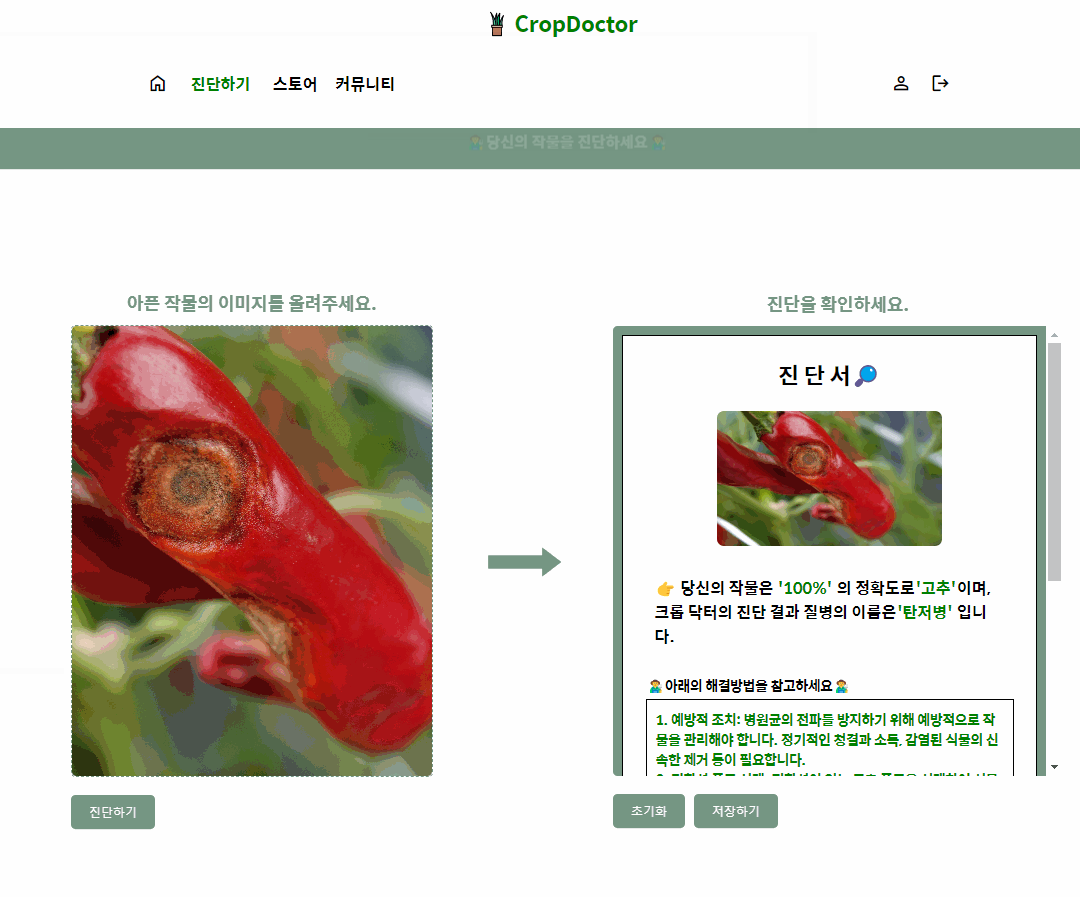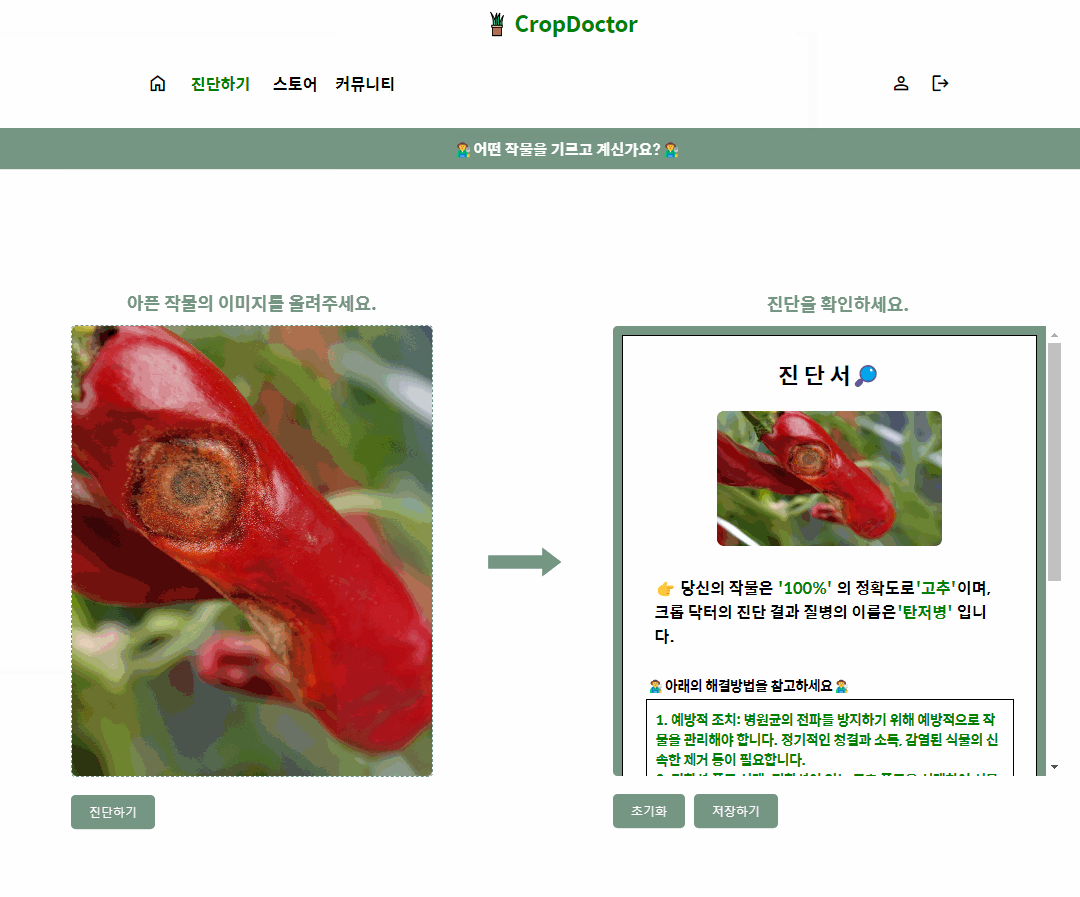
3.3 영양제 소개 페이지

3.4 커뮤니티 페이지

4. 마치며
이번 프로젝트에서는 nestjs, fastapi 등 기존에 사용해보지 않았던 새로운 기술들을 배워서 적용해 봤다는 점에서 의미가 큰 것 같다. 팀원 조정으로 총 4명이서 진행하였고, 5주라는 짧은 기간이었지만 완성도 있는 결과물을 낼 수 있었다.
백엔드 업무에서는 특히 데이터베이스 ERD를 설계했던 것이 기억에 남는다. 서비스에 필요한 테이블과 필드를 고민하고, 테이블 간의 관계 설정을 직접 함으로써 관계형 데이터베이스에 대한 이해도를 보다 높일 수 있었다.
인공지능 업무에서는 대용량 데이터 수집 부터 업로드, 환경에 맞는 전처리를 직접 설계하며 수행해 볼 수 있었고, 리눅스 환경에서 모델을 직접 학습해보는 전반적인 경험을 할 수 있었다. train 데이터셋과 비슷한 환경의 데이터인 validation 데이터셋은 예측을 잘 하는 듯 했지만, 외부에서 직접 가져온 데이터에 대해서는 잘 못하는 경우도 존재했는데, 물리적 자원이 부족하여 더 많은 실험을 해보지 못한 점이 아쉬웠다. 하지만, 프로젝트 환경 속에서 최대한 모델 성능을 높이고자 시도하기 위해 고민하는 시간 덕분에 배운 점이 많으며, 데이터 수집 - EDA - 전처리 - 모델학습 - 모델 평가- 모델 배포 까지의 전반적인 과정을 통해 데이터 과학과 머신 러닝에 대한 이해도가 보다 향상되었다. 데이터의 중요성, 전처리의 필요성, 모델의 선택과 평가, 그리고 실제 서비스에 모델을 배포하는 과정에서의 고려 사항을 배울 수 있었고, 문제 해결과 결과 도출을 위한 실험과 개선 과정에서의 시행착오와 학습을 통해 성장할 수 있었다. 또한 5주간 팀원들과 함께 협업하고 문제를 해결해 나가며 결과물을 완성시켜 뿌듯했고, 뜻깊은 경험이었다.
